近日,我所生物技术研究部生物分离分析新材料与新技术研究组(1809组)叶明亮研究员团队,联合大连理工大学董铭铭副教授、空军军医大学聂勇战教授等,开发了一款新型糖蛋白组学定量分析软件工具——GlyPep-Quant。该工具可显著降低大样本分析中糖肽定量的缺失值,提升位点特异性糖型的定量覆盖度,并应用于胃癌生物标志物的发现研究中。目前,GlyPep-Quant已作为重要工具集成于叶明亮团队前期发展的Glyco-Decipher糖肽解析软件,学术界可通过GitHub免费获取使用(https://github.com/DICP-1809)。
糖基化是蛋白质最复杂的翻译后修饰之一,其异常与癌症等多种疾病密切相关。然而,由于糖基化的高度异质性(同一蛋白质位点可能连接多种糖型)及低丰度特性,位点特异性糖基化的大规模定量分析长期面临谱图解析率低、定量缺失值多等难题,限制了其在疾病生物标志物发现中的应用。
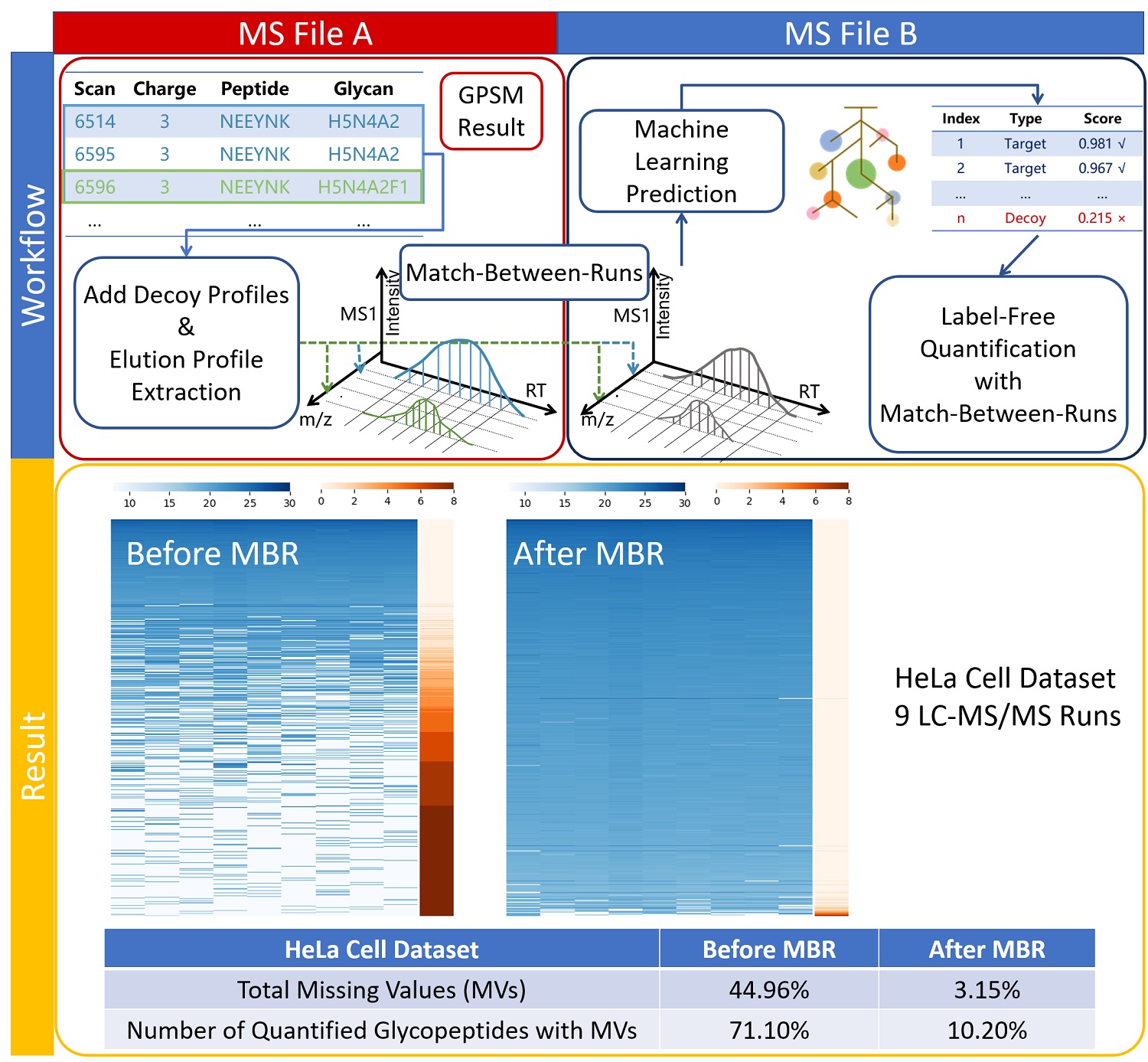
为了解决上述挑战,本工作提出了三个方面的解决方案:科研人员首先针对糖肽定量缺失值多的问题,发展了新型的基于机器学习的运行间匹配定量方法(match-between-runs,MBR)。研究发现,对基于Glyco-Decipher的鉴定结果进行定量时,缺失值比例约从70%降低为10%;与现有其他软件相比,GlyPep-Quant可多定量25.1%至178.9%的无缺失值的完整糖肽。其次,为了提高单个文件的鉴定灵敏度,科研人员提出了基于MS1特征库的虚拟匹配运行(library-based virtual MBR),利用已有大规模数据集的MS1特征信息构建“糖肽特征库”,并通过“库到新数据”的单向匹配策略,使新样本的位点特异性糖基化定量灵敏度较传统只依赖于MS2谱图鉴定的定量方法提升8倍以上,显著提高了单样本分析的覆盖深度。最后,科研人员提出了一种基于同一糖基化位点不同位点特异性糖型丰度比值的疾病标志物发现策略,有效消除个体蛋白表达差异及实验条件波动的干扰, 应用于分析胃癌大样本人血清数据集,发现了两个具有良好诊断性能的位点特异性糖型丰度比值,有望成为新型胃癌生物标志物。
叶明亮团队长期致力于蛋白质翻译后修饰分析新方法与配体靶蛋白质鉴定技术研究。在配体靶蛋白质鉴定方法方面,团队发展了具有自主知识产权的PELSA方法(Nat. Methods,2025),比国际同类的LiP-MS方法灵敏度高10倍以上。在蛋白质翻译后修饰方面,团队发展了第一个精氨酸二甲基化的化学富集方法(PNAS,2022),建立了能同时鉴定8种氨基酸甲基化的代谢标记方法(Nat. Commun.,2024),开发了用于O-GlcNAc糖肽富集的可逆酶促化学标记法 (Angew. Chem. Int. Ed.,2022)方法等。为适配大样本分析需求,团队开发了糖肽的自动化富集方法(Anal. Chem.,2021),构建了位点特异性糖型的高稳健分析系统(Adv. Sci.,2023);在糖肽谱图解析领域,开发了一款具有高灵敏度的N-糖肽质谱谱图解析新软件Glyco-Decipher(Nat. Commun.,2022),该软件利用不同糖肽的同一肽段骨架具有相似碎裂规律的特点,发展出基于“模式识别”的肽段序列鉴定新方法,从而提高了完整糖肽的鉴定灵敏度。
相关研究成果以“Library-based virtual match-between-runs quantification in GlyPep-Quant improves site-specific glycan identification”为题,于近日发表在《自然-通讯》(Nature Communications)上。该工作共同第一作者是1809组已毕业博士朱赫和博士后方正。上述研究得到国家重点研发计划、国家自然科学基金、我所创新基金等项目支持。(文/图 朱赫)