近期,我所生物技术研究部高分辨分离分析和代谢组学研究组(1808组)刘心昱研究员和许国旺研究员团队在代谢组学关键技术、方法研究中取得系列进展,提升了代谢组学分析的信息覆盖度、通量,以及数据挖掘与解析深度,为复杂生物体系小分子的精准表征及其在生物医学的应用提供了高效、可靠的技术手段。
人类健康受遗传、环境、菌群等多种因素及其相互作用的影响。以生物体内小分子化合物为研究对象的代谢组学技术在重大疾病的预防、诊断和治疗中正在发挥重要作用。然而,面对种类繁多、结构复杂、浓度跨度大的内源性代谢物及环境暴露物,当前代谢组学技术在全景式检测、数据处理及信息挖掘等方面面临诸多挑战。
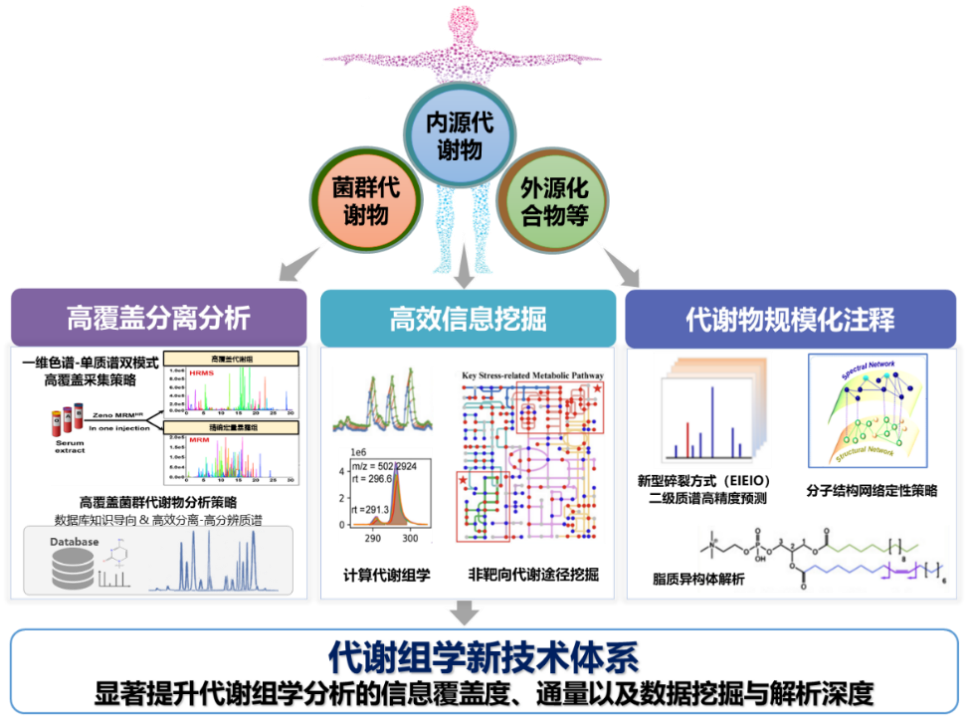
针对高覆盖分析需求,团队开发了基于一维色谱-单质谱双模式创新方法,实现了内源性代谢物与外源性暴露物的一体化同时分析,单次进样实现血中1,190个代谢物的半定量和210个暴露物的精准定量分析,提高了分析效率(Anal. Chem.,2025);发展了五氟苯基柱高覆盖检测结合数据库知识导向规模化注释的菌群相关代谢物系统性分析策略,拓宽了检测范围,单次进样可分析菌群相关的1,250种代谢物,为深入研究肠道微生物与宿主相互作用提供了有力支撑(Anal. Chem.,2024);开发了基于花状异质结纳米新材料的激光解吸电离质谱法(Anal. Chem.,2025),以及基于惯性微流控芯片的单细胞质谱法(Anal. Chem.,2024),有望在肺癌标志物发现及巨噬细胞极化的研究中发挥积极作用。
在代谢组学大数据处理方面,团队提出了适用于大规模样本非靶向质谱分析特征检测和匹配的MetCohort新方法,降低了检测的假阳性和假阴性,提升了大规模样本数据匹配的准确性,为后续的化合物鉴定和统计分析提供了保障(Anal. Chem.,2025);开发了基于化学分类驱动的网络分析揭示关键代谢途径的新方法,有效挖掘非靶向代谢组学中隐藏的代谢物结构信息,大大提高了组学数据利用率,为关键途径的限定提供了新方法(Anal. Chem.,2024)。
非靶向代谢组数据的注释一直是代谢组学发展的一个瓶颈。团队研发了系列基于结构大数据的分子网络注释新技术:将基于分子结构的分子网络方法(Anal. Chem.,2023)与新型有机物离子电子碰撞激发(EIEIO)技术(Anal. Chem.,2024)、新型质谱采集技术Orbitrap Astral(Anal. Chem.,2025)结合,进一步提升了注释的覆盖度与解析深度;研发的基于EIEIO的脂质sn-和C=C位置的精细结构注释方法,突破脂质精细结构解析的技术瓶颈,提升了复杂生物样本中脂质异构体的注释能力(Anal. Chem.,2025);提出了一种新型质谱碎片预测方法,最大程度保留化学结构信息,实现了小分子高分辨二级质谱的高精度预测(Anal. Chem.,2024)。
该团队在上述《分析化学》(Analytical Chemistry)上发表的关于代谢组学分析新技术的系列研究进展,提升了对代谢组的分析覆盖度、数据挖掘能力和规模化注释能力,突破了现有方法对质谱库的依赖。随着这些新技术、新方法的不断推广应用,有望在重大疾病的诊疗、药物研发、公共健康监测等诸多领域发挥积极作用。(文/图 路鑫、石先哲)